Primeiros Passos com tidyr e
dplyr
Lineu Alberto Cavazani de Freitas
1 Formatação e Tratamento de Dados
Vamos imaginar o processo de análise de um conjunto de dados, você precisa:
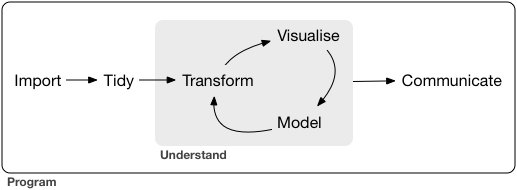
Cada uma delas consiste em:
- Importar os dados brutos para o software de análise.
- Arrumar os dados, isto é, fazer com que eles fiquem em um formato adequado para análise.
- Transformar os dados, isto é, criar novas variáveis com base nas que foram coletadas, obter resumos numéricos das variáveis de interesse, etc.
- Visualizar os dados, verificar o comportamento das variáveis e como elas interagem entre si fazendo uso de recursos gráficos. Leia o post Primeiros Passos com ggplot2 e entenda mais esta etapa.
- Modelar os dados, isto é, aplicar técnicas que vão além da visualização e permitam compreender os dados e aprender com eles.
- Comunicar os resultados.
Arrumar e Transformar os dados podem se tornar os piores pesadelos de estatísticos, cientistas de dados e aficcionados por análise. Estas duas etapas são de suma importância para uma análise consistente de dados e podem ser consideradas as mais demoradas, trabalhosas e desgastantes tarefas no processo de análise. Além disso são, sem dúvidas, etapas extremamente negligenciadas nos cursos de graduação e extremamente exigidas no mercado.
Existem diversos pacotes que permitem arrumar e transformar os dados, mas dois em especial merecem atenção: o tidyr e o dplyr. Estes pacotes são 2 componentes dos 8 pacotes básicos do tidyverse, se você nunca ouviu falar de tidyverse, trata-se de uma reimplementação das principais funcionalidades do R em um conjunto de 8 pacotes e uma série de vários outros secundários construídos para trabalhar em conjunto.
A vantagem de se trabalhar com o tidyverse é que toda a sintaxe e filosofia das diferentes etapas de análise são centralizadas e unificadas e, além disso, há um pacote específico para cada tarefa do processo de análise de dados. Um bom material para se inteirar a respeito do tidyverse é a série de aulas Manipulação e Visualização de Dados - A abordagem tidyverse do professor Walmes M. Zeviani e o material do Minicurso R 2019 ofertado pelo PET-Estatística UFPR.
Mas voltando ao que interessa, nosso foco neste post é apresentar o objetivo e principais funcionalidades do tidyr (para arrumação) e o dplyr (para transformação).

Para instalação dos pacotes basta utilizar a função
install.packages(), da seguinte forma:
install.packages('tidyr')
install.packages('dplyr')Ou ainda:
install.packages('tidyverse')Desta forma todos os pacotes pertencentes ao tidyverse serão instalados.
2 tidyr
Antes de dar início à exploração do tidyr vamos definir o conceito de dado arrumado ou tidy data. Um conjunto de dados considerado arrumado é aquele em que cada coluna representa uma variável, cada linha representa uma observação e cada célula representa o valor observado da observação i na variável j:

Isto posto, vamos às principais funcionalidades do
tidyr. Este pacote dispõe de funções para:
- Empilhar/desempilhar os dados.
- Separar/unir caracteres.
- Tratar dados ausenstes (NA’s).
- etc.
2.1 Empilhar/desempilhar os dados
Considere o seguinte conjunto de dados:
td <- tibble(
id = rep(1:50),
jan = rnorm(50, rpois(1,(runif(1)*10))),
fev = rnorm(50, rpois(1,(runif(1)*10))),
mar = rnorm(50, rpois(1,(runif(1)*10))),
abr = rnorm(50, rpois(1,(runif(1)*10))),
mai = rnorm(50, rpois(1,(runif(1)*10))),
jun = rnorm(50, rpois(1,(runif(1)*10))),
jul = rnorm(50, rpois(1,(runif(1)*10))),
ago = rnorm(50, rpois(1,(runif(1)*10))),
set = rnorm(50, rpois(1,(runif(1)*10))),
out = rnorm(50, rpois(1,(runif(1)*10))),
nov = rnorm(50, rpois(1,(runif(1)*10))),
dez = rnorm(50, rpois(1,(runif(1)*10))))
td## # A tibble: 50 × 13
## id jan fev mar abr mai jun jul ago set out nov
## <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 1.86 0.869 2.83 7.52 11.0 2.51 10.8 2.52 4.50 8.27 14.3
## 2 2 1.30 -0.129 0.434 7.88 8.38 2.16 10.3 0.541 5.54 8.63 14.6
## 3 3 3.35 1.16 1.63 5.71 9.81 0.890 9.95 1.75 5.93 8.39 14.9
## 4 4 5.58 1.30 0.736 6.01 9.25 0.719 10.0 -1.12 5.82 7.52 15.3
## 5 5 3.41 1.57 0.929 3.92 9.18 1.37 10.9 -0.172 7.23 8.11 15.4
## 6 6 5.68 1.43 0.728 6.40 9.67 0.359 10.1 0.503 4.62 7.81 15.0
## 7 7 4.87 1.64 2.49 6.82 10.7 1.48 10.4 -1.37 4.16 6.67 14.7
## 8 8 4.13 1.97 0.693 4.67 8.82 2.06 10.7 -0.178 4.96 6.89 14.9
## 9 9 1.75 1.97 -0.162 6.88 8.84 1.36 11.4 -0.417 5.12 7.88 12.8
## 10 10 4.72 1.34 3.62 5.84 8.90 2.72 10.6 -0.725 3.82 7.47 17.0
## # … with 40 more rows, and 1 more variable: dez <dbl>Use a imaginação, finja que cada valor de id é uma pessoa, cada coluna um mês e cada célula o resultado de um exame qualquer. Este é o típico caso de dados no formato largo, ou seja, a linha representa uma observação mas note que as colunas representam a mesma variável: o mês. No formato tido como ideal deveria haver uma coluna id e uma coluna mês e não 12 colunas de uma mesma variável.
Para forçar os dados a assumirem o formato longo basta utilizar a
função gather, nesta função você fornece o nome de duas
colunas a serem criadas: a key e a value. Além disso deve-se informar as
colunas que se tem interesse em empilhar.
td_gat <- td %>% gather(key = 'mes', value = 'valor', jan:dez)
td_gat## # A tibble: 600 × 3
## id mes valor
## <int> <chr> <dbl>
## 1 1 jan 1.86
## 2 2 jan 1.30
## 3 3 jan 3.35
## 4 4 jan 5.58
## 5 5 jan 3.41
## 6 6 jan 5.68
## 7 7 jan 4.87
## 8 8 jan 4.13
## 9 9 jan 1.75
## 10 10 jan 4.72
## # … with 590 more rowsPara fazer com que os dados retornem ao formato original basta
utilizar a função spread:
td_gat %>% spread(key=mes, value = valor)## # A tibble: 50 × 13
## id abr ago dez fev jan jul jun mai mar nov out
## <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 7.52 2.52 1.05 0.869 1.86 10.8 2.51 11.0 2.83 14.3 8.27
## 2 2 7.88 0.541 0.747 -0.129 1.30 10.3 2.16 8.38 0.434 14.6 8.63
## 3 3 5.71 1.75 2.72 1.16 3.35 9.95 0.890 9.81 1.63 14.9 8.39
## 4 4 6.01 -1.12 2.06 1.30 5.58 10.0 0.719 9.25 0.736 15.3 7.52
## 5 5 3.92 -0.172 1.49 1.57 3.41 10.9 1.37 9.18 0.929 15.4 8.11
## 6 6 6.40 0.503 2.75 1.43 5.68 10.1 0.359 9.67 0.728 15.0 7.81
## 7 7 6.82 -1.37 -0.214 1.64 4.87 10.4 1.48 10.7 2.49 14.7 6.67
## 8 8 4.67 -0.178 1.72 1.97 4.13 10.7 2.06 8.82 0.693 14.9 6.89
## 9 9 6.88 -0.417 -0.283 1.97 1.75 11.4 1.36 8.84 -0.162 12.8 7.88
## 10 10 5.84 -0.725 2.08 1.34 4.72 10.6 2.72 8.90 3.62 17.0 7.47
## # … with 40 more rows, and 1 more variable: set <dbl>Mas note a diferença, o fragmento anterior fez com que as colunas fossem colocadas em ordem alfabética.
2.2 Separar/unir caracteres.
Imagine um exemplo simples, você coletou dia, mês e ano de nascimento
de uma série de indivíduos mas cada um destes valores está em uma célula
e você quer que isto fique numa única célula. Este é um caso clássico em
que a função unite se mostra útil:
dma <-
tibble(dia = c(rpois(10, 10)),
mes = c(01,02,03,04,05,06,07,08,09,10),
ano = c(1990,1994,1996,2001,2005,1955,1998,1999,2005,1955))
dma## # A tibble: 10 × 3
## dia mes ano
## <int> <dbl> <dbl>
## 1 8 1 1990
## 2 11 2 1994
## 3 9 3 1996
## 4 6 4 2001
## 5 9 5 2005
## 6 7 6 1955
## 7 9 7 1998
## 8 5 8 1999
## 9 10 9 2005
## 10 10 10 1955dma2 <- dma %>% unite(col = 'data', sep = '-')
dma2## # A tibble: 10 × 1
## data
## <chr>
## 1 8-1-1990
## 2 11-2-1994
## 3 9-3-1996
## 4 6-4-2001
## 5 9-5-2005
## 6 7-6-1955
## 7 9-7-1998
## 8 5-8-1999
## 9 10-9-2005
## 10 10-10-1955Para fazer com que os dados retomem o formato original uma candidata
é a função separate:
dma2 %>% separate(col = 'data',
into = c('dia', 'mes', 'ano'),
sep = '-')## # A tibble: 10 × 3
## dia mes ano
## <chr> <chr> <chr>
## 1 8 1 1990
## 2 11 2 1994
## 3 9 3 1996
## 4 6 4 2001
## 5 9 5 2005
## 6 7 6 1955
## 7 9 7 1998
## 8 5 8 1999
## 9 10 9 2005
## 10 10 10 1955Já que entramos no assunto datas, recomendo a leitura do post Resolvendo problemas com datas e horas utilizando o pacote Lubridate
2.3 Tratar dados ausenstes (NA’s).
Dados ausentes são um assunto delicado. Há quem defenda a completa retirada de linhas com valores faltantes, há também quem defenda que estes valores tem algo a contar e devem permanecer na base. Fato é que em determinados métodos não há a possibilidade de trabalhar com uma base em que haja dados ausentes, como é o caso de modelos de regressão. Nestes casos nos restam algumas alternativas: retirar linhas em que haja algum NA, substituir NA’s por alguma outra quantidade ou utilizar algum método de imputação.
Não é o foco deste post discutir qual é mais recomendada ou coerente,
mas sim mostrar as possibilidades com o tidyr, e são elas:
retirar as linhas com NA com a função drop_na() ou
substituir os NA’s oir algum outro valor com a função
replace_na():
NAS <- tibble(col1 = 1:10,
col2 = c(1,2,NA,4,NA,6,7,NA,9,10))
NAS## # A tibble: 10 × 2
## col1 col2
## <int> <dbl>
## 1 1 1
## 2 2 2
## 3 3 NA
## 4 4 4
## 5 5 NA
## 6 6 6
## 7 7 7
## 8 8 NA
## 9 9 9
## 10 10 10NAS %>% drop_na()## # A tibble: 7 × 2
## col1 col2
## <int> <dbl>
## 1 1 1
## 2 2 2
## 3 4 4
## 4 6 6
## 5 7 7
## 6 9 9
## 7 10 10NAS %>% replace_na(list(col2 = 100000))## # A tibble: 10 × 2
## col1 col2
## <int> <dbl>
## 1 1 1
## 2 2 2
## 3 3 100000
## 4 4 4
## 5 5 100000
## 6 6 6
## 7 7 7
## 8 8 100000
## 9 9 9
## 10 10 103 dplyr
O dplyr dispõe de funções para transformação do conjunto
de dados. Isso envolve: filtrar linhas, selecionar colunas, ordenar a
base, criar/modificar colunas, agrupar a base, sumarizar e cruzar
diferentes bases. Considere o conjunto de dados swiss.
swiss$names <- rownames(swiss)
dados <- swiss %>% as_tibble()
summary(dados)## Fertility Agriculture Examination Education
## Min. :35.00 Min. : 1.20 Min. : 3.00 Min. : 1.00
## 1st Qu.:64.70 1st Qu.:35.90 1st Qu.:12.00 1st Qu.: 6.00
## Median :70.40 Median :54.10 Median :16.00 Median : 8.00
## Mean :70.14 Mean :50.66 Mean :16.49 Mean :10.98
## 3rd Qu.:78.45 3rd Qu.:67.65 3rd Qu.:22.00 3rd Qu.:12.00
## Max. :92.50 Max. :89.70 Max. :37.00 Max. :53.00
## Catholic Infant.Mortality names
## Min. : 2.150 Min. :10.80 Length:47
## 1st Qu.: 5.195 1st Qu.:18.15 Class :character
## Median : 15.140 Median :20.00 Mode :character
## Mean : 41.144 Mean :19.94
## 3rd Qu.: 93.125 3rd Qu.:21.70
## Max. :100.000 Max. :26.603.1 Filtrar linhas
A função filter funciona como um filtro de uma planilha Excel, você
escolhe uma condição e o output serão apenas aquelas linhas em que a
condição seja satisfeita. Seguem alguns exemplos utilizando a função
filter():
- Filtro comum: filtre as linhas em que apenas esta condição seja satisfeita.
- Filtro com condição &: filtre as linhas em que duas condições que sejam satisfeitas simultâneamente.
- Filtro com condição |: filtre as linhas em que uma das duas condições seja atendida.
- Filtro com condição %in%: filtre as linhas em que haja observações iguais às pertencenter ao vetor.
3.1.1 Filtro comum
filtro1 <- dados %>% filter(Fertility > 70)
nrow(dados) - nrow(filtro1)## [1] 2323 observações não atendem à restrição.
3.1.2 Filtro com condição &
filtro2 <- dados %>% filter(Examination > 12 & Agriculture > 30)
nrow(dados) - nrow(filtro2)## [1] 2424 observações não atendem à restrição.
3.1.3 Filtro com condição |
filtro3 <- dados %>% filter(Examination > 12 | Agriculture < 30)
nrow(dados) - nrow(filtro3)## [1] 1414 observações não atendem à restrição.
3.1.4 Filtro com condição %in%
filtro4 <- dados %>% filter(names %in% c("Courtelary", "Delemont", "Franches-Mnt"))
nrow(dados) - nrow(filtro4)## [1] 4444 observações não atendem à restrição.
3.2 Selecionar colunas
A função select() é utilizada para selecionar colunas de
um conjunto de dados, com esta função é possível:
- Selecionar colunas pelo nome.
- Selecionar colunas que iniciam com alguma letra ou sílaba específica.
- Selecionar intervalos entre colunas.
- Selecionar todas com exceção de algumas.
3.2.1 Selecionando colunas pelo nome
select1 <- dados %>% select(names, Education)
names(dados)## [1] "Fertility" "Agriculture" "Examination" "Education"
## [5] "Catholic" "Infant.Mortality" "names"names(select1)## [1] "names" "Education"3.2.2 Selecionando colunas que iniciam com
select2 <- dados %>% select(starts_with("e"))
names(dados)## [1] "Fertility" "Agriculture" "Examination" "Education"
## [5] "Catholic" "Infant.Mortality" "names"names(select2)## [1] "Examination" "Education"3.2.3 Selecionando intervalo de colunas
select3 <- dados %>% select(Fertility, Agriculture:Education)
names(dados)## [1] "Fertility" "Agriculture" "Examination" "Education"
## [5] "Catholic" "Infant.Mortality" "names"names(select3)## [1] "Fertility" "Agriculture" "Examination" "Education"3.2.4 Selecionando todas as colunas exceto
select4 <- dados %>% select(-Fertility, -names)
names(dados)## [1] "Fertility" "Agriculture" "Examination" "Education"
## [5] "Catholic" "Infant.Mortality" "names"names(select4)## [1] "Agriculture" "Examination" "Education" "Catholic"
## [5] "Infant.Mortality"3.3 Ordenar a base
Para ordenação com base em uma coluna o dplyr dispõe da
função arrange():
df <- tibble(letras = letters,
valor = rnorm(length(letters)))
df## # A tibble: 26 × 2
## letras valor
## <chr> <dbl>
## 1 a -0.630
## 2 b 0.608
## 3 c -0.944
## 4 d -1.16
## 5 e -0.0887
## 6 f 0.541
## 7 g -1.47
## 8 h 1.41
## 9 i -1.33
## 10 j -1.32
## # … with 16 more rowsdf %>% arrange(desc(letras))## # A tibble: 26 × 2
## letras valor
## <chr> <dbl>
## 1 z -0.245
## 2 y -0.841
## 3 x -0.729
## 4 w 0.416
## 5 v -1.92
## 6 u -0.299
## 7 t 0.522
## 8 s 0.599
## 9 r -0.395
## 10 q -0.146
## # … with 16 more rowsdf %>% arrange(letras)## # A tibble: 26 × 2
## letras valor
## <chr> <dbl>
## 1 a -0.630
## 2 b 0.608
## 3 c -0.944
## 4 d -1.16
## 5 e -0.0887
## 6 f 0.541
## 7 g -1.47
## 8 h 1.41
## 9 i -1.33
## 10 j -1.32
## # … with 16 more rows3.4 Criar/modificar colunas
A criação e modificação de colunas pode ser feita com a função
mutate(), com ela é possível:
- Sobrescreve uma coluna.
- Criar uma nova coluna.
- Criar diversas colunas no mesmo mutate.
df2 <- tibble(col1 = rnorm(50), col2 = rpois(50, 1))
df2## # A tibble: 50 × 2
## col1 col2
## <dbl> <int>
## 1 0.588 1
## 2 -1.14 2
## 3 0.940 0
## 4 -1.13 0
## 5 -0.283 1
## 6 0.493 0
## 7 -1.64 0
## 8 0.724 0
## 9 1.79 2
## 10 -0.646 1
## # … with 40 more rows3.4.1 Sobrescreve uma coluna
mutate1 <- df2 %>% mutate(col2 = letters[1:10] %>% rep(5))
df2[,2]## # A tibble: 50 × 1
## col2
## <int>
## 1 1
## 2 2
## 3 0
## 4 0
## 5 1
## 6 0
## 7 0
## 8 0
## 9 2
## 10 1
## # … with 40 more rowsmutate1[,2]## # A tibble: 50 × 1
## col2
## <chr>
## 1 a
## 2 b
## 3 c
## 4 d
## 5 e
## 6 f
## 7 g
## 8 h
## 9 i
## 10 j
## # … with 40 more rows3.4.2 Criar uma nova coluna
mutate2 <- df2 %>% mutate(col3 = 1:nrow(df2))
df2## # A tibble: 50 × 2
## col1 col2
## <dbl> <int>
## 1 0.588 1
## 2 -1.14 2
## 3 0.940 0
## 4 -1.13 0
## 5 -0.283 1
## 6 0.493 0
## 7 -1.64 0
## 8 0.724 0
## 9 1.79 2
## 10 -0.646 1
## # … with 40 more rowsmutate2## # A tibble: 50 × 3
## col1 col2 col3
## <dbl> <int> <int>
## 1 0.588 1 1
## 2 -1.14 2 2
## 3 0.940 0 3
## 4 -1.13 0 4
## 5 -0.283 1 5
## 6 0.493 0 6
## 7 -1.64 0 7
## 8 0.724 0 8
## 9 1.79 2 9
## 10 -0.646 1 10
## # … with 40 more rows3.4.3 Criar diversas colunas no mesmo mutate.
mutate3 <- df2 %>% mutate( col3 = col1 + col2,
col4 = 1:nrow(df2),
col5 = col1 * 10)
df2## # A tibble: 50 × 2
## col1 col2
## <dbl> <int>
## 1 0.588 1
## 2 -1.14 2
## 3 0.940 0
## 4 -1.13 0
## 5 -0.283 1
## 6 0.493 0
## 7 -1.64 0
## 8 0.724 0
## 9 1.79 2
## 10 -0.646 1
## # … with 40 more rowsmutate3## # A tibble: 50 × 5
## col1 col2 col3 col4 col5
## <dbl> <int> <dbl> <int> <dbl>
## 1 0.588 1 1.59 1 5.88
## 2 -1.14 2 0.858 2 -11.4
## 3 0.940 0 0.940 3 9.40
## 4 -1.13 0 -1.13 4 -11.3
## 5 -0.283 1 0.717 5 -2.83
## 6 0.493 0 0.493 6 4.93
## 7 -1.64 0 -1.64 7 -16.4
## 8 0.724 0 0.724 8 7.24
## 9 1.79 2 3.79 9 17.9
## 10 -0.646 1 0.354 10 -6.46
## # … with 40 more rows3.5 Agrupar e sumarizar
A função summarise() é a responsável por gerar resumos
numéricos, o output desta função é um tibble em que você nomeia a coluna
e especifica que medida será printada. Considere novamente o conjunto de
dados swiss:
swiss$names <- rownames(swiss)
dados <- swiss %>% as_tibble()
summary(dados)## Fertility Agriculture Examination Education
## Min. :35.00 Min. : 1.20 Min. : 3.00 Min. : 1.00
## 1st Qu.:64.70 1st Qu.:35.90 1st Qu.:12.00 1st Qu.: 6.00
## Median :70.40 Median :54.10 Median :16.00 Median : 8.00
## Mean :70.14 Mean :50.66 Mean :16.49 Mean :10.98
## 3rd Qu.:78.45 3rd Qu.:67.65 3rd Qu.:22.00 3rd Qu.:12.00
## Max. :92.50 Max. :89.70 Max. :37.00 Max. :53.00
## Catholic Infant.Mortality names
## Min. : 2.150 Min. :10.80 Length:47
## 1st Qu.: 5.195 1st Qu.:18.15 Class :character
## Median : 15.140 Median :20.00 Mode :character
## Mean : 41.144 Mean :19.94
## 3rd Qu.: 93.125 3rd Qu.:21.70
## Max. :100.000 Max. :26.603.5.1 Obtendo uma única medida descritiva
dados %>%
summarise(mean_fert = mean(Fertility))## # A tibble: 1 × 1
## mean_fert
## <dbl>
## 1 70.13.5.2 Obtendo mais de uma medida descritiva
dados %>%
summarise(
media_fert = mean(Fertility),
mediana_fert = median(Fertility))## # A tibble: 1 × 2
## media_fert mediana_fert
## <dbl> <dbl>
## 1 70.1 70.43.5.3 Obtendo medidas descritivas de diferentes variáveis
dados %>%
summarise(
media_fert = mean(Fertility),
mediana_agr = median(Agriculture),
sd_cat = sd(Catholic))## # A tibble: 1 × 3
## media_fert mediana_agr sd_cat
## <dbl> <dbl> <dbl>
## 1 70.1 54.1 41.73.5.4 Obtendo medidas descritivas por grupo
O dplyr possui a função group_by(). Esta
função, quando combinada ao summarise() permite obter
medidas descritivas por grupo. Imagine que você tem no seu conjunto de
dados uma variável categórica de 3 níveis e uma variável contínua,
combinando group_by() e summarise() você pode
obter a média da variável contínua para cada nível da variável
categórica sem muito esforço, veja:
dados <- tibble(fator = rep(c('a','b','c'), 5),
num = rnorm(length(fator), 15,5))
dados## # A tibble: 15 × 2
## fator num
## <chr> <dbl>
## 1 a 6.90
## 2 b 13.1
## 3 c 19.1
## 4 a 14.4
## 5 b 17.6
## 6 c 17.2
## 7 a 8.95
## 8 b 4.01
## 9 c 18.1
## 10 a 8.60
## 11 b 18.7
## 12 c -0.0939
## 13 a 16.1
## 14 b 26.2
## 15 c 13.6dados %>%
group_by(fator) %>%
summarise(media_grupo = mean(num))## # A tibble: 3 × 2
## fator media_grupo
## <chr> <dbl>
## 1 a 11.0
## 2 b 15.9
## 3 c 13.63.6 Amostrar
Em alguns casos, para aplicação de métodos específicos, pode ser que
seja interesse trabalhar com amostras dos dados. O dplyr
possui duas funções para reamostragem: a sample_n() e a
sample_frac(). Na primeira você informa quantas linhas do
conjunto de dados original você deseja e na segunda a proporção de
linhas do conjunto de dados original:
dados ## # A tibble: 15 × 2
## fator num
## <chr> <dbl>
## 1 a 6.90
## 2 b 13.1
## 3 c 19.1
## 4 a 14.4
## 5 b 17.6
## 6 c 17.2
## 7 a 8.95
## 8 b 4.01
## 9 c 18.1
## 10 a 8.60
## 11 b 18.7
## 12 c -0.0939
## 13 a 16.1
## 14 b 26.2
## 15 c 13.6dados %>% sample_n(5)## # A tibble: 5 × 2
## fator num
## <chr> <dbl>
## 1 b 4.01
## 2 b 17.6
## 3 a 14.4
## 4 b 26.2
## 5 c 17.2dados %>% sample_frac(0.1)## # A tibble: 2 × 2
## fator num
## <chr> <dbl>
## 1 a 8.95
## 2 c -0.09393.7 Cruzamentos
As funções do tipo join servem para cruzar bases por uma coluna chave, as possíveis alternativas de cruzamento são:
inner_join().full_join().left_join().right_join().
A inner_join() gera um novo conjunto de dados apenas
para os casos em que a chave é verificada nos dois arquivos.
A full_join() une todas as linhas e todas as colunas,
independente da chave ocorrer nos dois arquivos. Nos casos em que não há
correspondência é gerado NA.
Na left_join() todas as chaves do primeiro arquivo são
mantidas e acrescenta-se valores nos casos em que há compatibilidade de
chave. As chaves que são observadas apenas no segundo conjunto de dados
são descartados.
No right_join() todas as chaves do segundo arquivo são
mantidas e acrescenta-se valores nos casos em que há compatibilidade de
chave. As chaves que são observadas apenas no primeiro conjunto de dados
são descartados.

Para aplicação, considere os 2 seguintes conjuntos de dados:
tb1 <- tibble(codigo = c("001", "002", "003", "004", "005",
"006", "007", "008", "009", "010"),
ocorrencia = rbinom(10,1, prob = 0.5),
n = rpois(10,2))
tb2 <- tibble(code = c("001", "022", "003", "004", "005",
"000", "007", "018", "079", "010"),
year = c(2010, 2011, 2015, 1999, 2000,
1996, 1998, 1955, 1971, 2001))
tb1## # A tibble: 10 × 3
## codigo ocorrencia n
## <chr> <int> <int>
## 1 001 1 4
## 2 002 1 2
## 3 003 0 7
## 4 004 1 3
## 5 005 0 3
## 6 006 1 3
## 7 007 0 2
## 8 008 1 3
## 9 009 1 1
## 10 010 0 2tb2## # A tibble: 10 × 2
## code year
## <chr> <dbl>
## 1 001 2010
## 2 022 2011
## 3 003 2015
## 4 004 1999
## 5 005 2000
## 6 000 1996
## 7 007 1998
## 8 018 1955
## 9 079 1971
## 10 010 20013.7.1 inner_join()
inner_join(tb1, tb2, by = c("codigo"="code"))## # A tibble: 6 × 4
## codigo ocorrencia n year
## <chr> <int> <int> <dbl>
## 1 001 1 4 2010
## 2 003 0 7 2015
## 3 004 1 3 1999
## 4 005 0 3 2000
## 5 007 0 2 1998
## 6 010 0 2 20013.7.2 full_join()
full_join(tb1, tb2, by = c("codigo"="code"))## # A tibble: 14 × 4
## codigo ocorrencia n year
## <chr> <int> <int> <dbl>
## 1 001 1 4 2010
## 2 002 1 2 NA
## 3 003 0 7 2015
## 4 004 1 3 1999
## 5 005 0 3 2000
## 6 006 1 3 NA
## 7 007 0 2 1998
## 8 008 1 3 NA
## 9 009 1 1 NA
## 10 010 0 2 2001
## 11 022 NA NA 2011
## 12 000 NA NA 1996
## 13 018 NA NA 1955
## 14 079 NA NA 19713.7.3 right_join()
right_join(tb1, tb2, by = c("codigo"="code"))## # A tibble: 10 × 4
## codigo ocorrencia n year
## <chr> <int> <int> <dbl>
## 1 001 1 4 2010
## 2 003 0 7 2015
## 3 004 1 3 1999
## 4 005 0 3 2000
## 5 007 0 2 1998
## 6 010 0 2 2001
## 7 022 NA NA 2011
## 8 000 NA NA 1996
## 9 018 NA NA 1955
## 10 079 NA NA 19713.7.4 left_join()
left_join(tb1, tb2, by = c("codigo"="code"))## # A tibble: 10 × 4
## codigo ocorrencia n year
## <chr> <int> <int> <dbl>
## 1 001 1 4 2010
## 2 002 1 2 NA
## 3 003 0 7 2015
## 4 004 1 3 1999
## 5 005 0 3 2000
## 6 006 1 3 NA
## 7 007 0 2 1998
## 8 008 1 3 NA
## 9 009 1 1 NA
## 10 010 0 2 20014 Considerações finais
Neste post exploramos dois pacotes tidyverse: o
tidyr e o dplyr. Estes pacotes são
extremamente úteis dada a necessidade de ferramentas para manipulação
consistente de dados, a principal vantagem destes pacotes é a sintaxe e
nome das funções. De forma geral as estas ferramentas possuem funções e
argumentos com nomes consideravelmente mais intuitivos que pacotes
antigos e já consolidados dentre usuários de R e se mostram com
importantes funcionalidades a serem estudados por aqueles interessados
por análise de dados bem como os demais pacotes pertencentes ao
tidyverse.